(ISSN 1430-6972)
IP-GIPT DAS=14.11.2002 Internet-Erstausgabe, letzte Änderung: 19.01.20
Impressum: Diplom-PsychologInnen Irmgard Rathsmann-Sponsel und Dr. phil. Rudolf Sponsel
Stubenlohstr. 20 D-91052 Erlangen * Mail: sekretariat@sgipt.org
Anfang_ q50__Datenschutz_Überblick _ Relativ Aktuelles _ Rel. Beständiges _ Titelblatt _ Konzept _ Archiv _ Region _ Service iec-verlag _ _ Zitierung & Copyright
Willkommen in der Abteilung Wissenschaftstheorie, Methodologie und Statistisch-Mathematische Methoden in der Allgemeinen und Integrativen Psychologie, Psychodiagnostik und Psychotherapie, hier:(Entwicklungs- und Diskussionsversion)
30 Versuche mit zufällig normalverteilter Fehlerspannweite von 50% bezüglich der definierten wahren Werte des Quaders zur explorativen Untersuchung des Verhaltens der Eigenwerte und Faktoren
Materialien und Dokumente zur Kritik der Handhabung der Faktorenanalyse.
von Rudolf Sponsel, Erlangen
Überblicks- und Verteilerseite
zu diesen Simulations-Versuchen * Querverweise
- Beschreibung dieser Versuchsserie: Was wurde gemacht?
- Vollständiges Beispiel aus dieser Versuchsserie, hier F50v15
- Drei Beispielgraphen zur Meßwertsimulation der Serie 1-30
- Urdatenliste dieses Versuchsserien Beispiels F50v15
- Standard-Matrixanalyse der Korrelationsmatrix des Beispiels F50v15
- Die drei Hauptfaktoren und Rückrechnung der Reproduktionsmatrix des Beispiels mit Residualanalysen der Original-Korrelationmatriv mit aus allen 8 und 3-Faktoren rückgerechneten Koeffizienten des Beispiels F50v15
- Reproduktionen der Originalkorrelationsmatrix aus den drei konstituierenden Faktoren des Quaders dieser 30 Fehlerversuche F50v1-30
- Eigenwertstatistik dieser dreißig Fehlerversuche mit 50% Spannweite (WW+- 25%)
- Zusammenfassung/ Abstract Ergebnisse F50v1-30
Beschreibung dieser Versuchsserie: Was wurde gemacht ?
Ausgehend von den wahren Werten der
Ausgangsbasis wurden für jeden der 800 Werte - 100 Quader-
'ProbandInnen' mit insgesamt 8, davon drei unabhängigen und 5 linear
abhängigen Variablen - mit dem Zufallsgenerator
von Jörn Wilms zufällig normalverteilte Fehlerwerte für
die Fehlerspannweite 50% (WW +- 25%) für die 30 verschiedenen Ziehungen
erzeugt [drei Beispielgraphen hier] aus denen eine Versuchsserie besteht.
Dann wurden die 30 Produkt-Moment-Korrelationsmatrizen berechnet. Anschließend
wurden die 30 Korrelationsmatrizen einer
Standardmatrix-Analyse
(Sponsel
1994) unterzogen und dabei die Eigenwerte und Eigenvektoren berechnet,
woraus dann die Faktoren nach der Hauptkomponentenmethode von Cooley
& Lohnes gewonnen wurden. Kollinearitäts- und rundungsbedingte
kleine negative Eigenwerte traten in dieser Serie nicht mehr auf. Anschließend
wurden aus den Faktoren, einmal aus allen 8, einmal aus den ersten 3 Faktoren
Reproduktionsmatrizen erstellt und die Mittelwerte und Standardabweichungen
der Abweichungen nach einer Residualanalysemethode
von der Originalkorrelationsmatrizen auf der Basis 17stelliger Genauigkeit
berechnet. Wie die Mathematik erwarten läßt, gelingt die Reproduktion
der Originalkorrelationsmatrix mit allen 8 Faktoren vollständig
und die aus den ersten drei Faktoren in Abhängigkeit von der Fehlerspanne
nur mehr oder minder angenähert, hier für die Fehlerspannweite
50% überraschend gut.
| Sinn dieser F%*30 Eigenwert-, Faktoren- und Residualanalysen ist, Aufschlüsse darüber zu erhalten, wie sich die Eigenwerte, Faktorenanalysen und Reproduktionsgüten in Abhängigkeit von den Fehlerspannweiten verhalten und entwickeln. |
Es wurde bewußt ein Quader gewählt, weil dieser unserer dreidimensionalen Anschauung sehr zugänglich ist und weil wir jeden Wert und jede Veränderung ganz genau kennen und dokumentieren können.
Drei Beispielgraphen zur Meßwertsimulation der Versuche 1-30
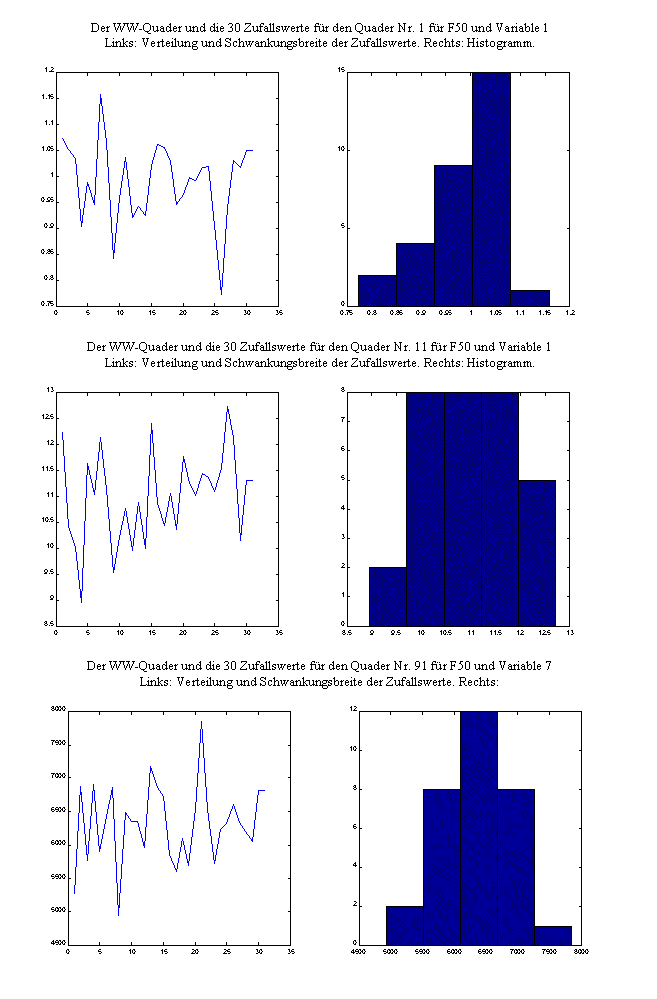
Anmerkung: Die Normalverteilung bei nur 30 Ziehungen und 50% Fehlerspannweite ist in nicht wenigen Fällen nur mit Phantasie und gutwilligem Wissen erkennbar, im letzten Beispiel Quader 91in Variable 7 allerdings sehr klar und schön.
Vollständiges Beispiel aus dieser Versuchsserie, hier F50v15
Hinweis: Aus Darstellungsgründen sind die Werte gerundet. Intern wurde mit 17stelliger Genauigkeit gerechnet. Die genauen Werte werden am Ende des Versuchs in einer CD-ROM im ASCII-Format angeboten.
Urdatenliste des Beispiels F50v15
Hinweis: Aus Darstellungsgründen sind die Werte gerundet. Intern wurde mit 17stelliger Genauigkeit gerechnet. Die genauen Werte werden am Ende des Versuchs in einer CD-ROM im ASCII-Format angeboten.
1
2 3
4 5
6 7
8
i\j: Länge Breite Höhe
Volum Oberfl L*B L*H
B*H
1 1.022 93.916 61.26
5086.1 11982 94 60
5619.7
2 2.2 67.1
71.6 9528.6 9332.7 128.48 159.61
4802.6
3 3.1 21.7
39.8 2444.7 1878.1 61.4
110.47 860.37
4 3.74 75.02
95.69 28577 14453 286.9
345.98 6126.3
5 5.7 74.9
8.2 3101.3 2297.7 510.98 40.65
718.95
6 6.05 99.77
72.38 39193 16365 587.35 420.26
7292.7
7 7.6 37
39.5 14368 5523.5 326.15 294.59
2095.9
8 7.4 19.3
73.7 9387.9 4822.3 149.25 649.97
1533.1
9 8.1 13.6
90.7 10528 3555 114.92
748.8 1077.5
10 9.1 58.9
36.8 22458 6815.3 595.56 398.24
1825.3
11 12.4 12.5
69.7 11249 3518.4 116.44 809.18
938.02
12 13.18 1.02 58.14
630.33 1439.5 13.7 700.13 49.79
13 13.22 40.26 78.22
37288 8363 547.35 1093.3 2899.2
14 13.1 29.9
19.3 8476.6 2928.1 548.29 284.28
558.02
15 14.26 70.5 31.8
41666 9748.5 1364.8 548.82 3101.6
16 16 19.7
31.5 8556.6 2532.7 298.88 488.05
523.82
17 16.07 94.66 55.38
91394 14810 1659.2 827.98 6061.1
18 17.2 71.4
51.6 72841 13254 1603.7
973.11 3699.5
19 17.9 74
37.3 67196 12962 1643.5
776.84 3407.6
20 22.5 79.9
37.1 57135 10796 1651.2
767.82 2969.7
21 21 46.1
58.8 49923 10315 1320.7
980.92 2511.1
22 21.2 84.9
19.1 35933 9378.3 1695.5 385.64
1561.1
23 23.4 35.2
60.7 51271 9896.5 971.34 1182.5
2069.6
24 24.2 77.4
71.3 126745 16213 1666.8 1893.5
4393.6
25 30.6 81.9
51.9 105502 13284 2294
1076.4 3584.2
26 28.9 74.4
37.8 85897 10278 1996.8
1103 2865.1
27 24.5 49.8
2.2 2496.7 2562.1 1322.6 54.2
105.54
28 32.06 56.18 42.32
65895 9271.4 1348.1 1087.4 2180.8
29 30.8 33.3
50.4 50042 6304.1 971.29 1407.4
1613.5
30 27.2 36.9
72.1 89988 14096 981.67
2330.7 3112.5
31 29.7 86.7
71 174796 19643 2483.4
2412.5 6405.9
32 29.7 58.1
61.9 146428 18122 1818
2114.5 4816.8
33 35.8 48.1
10.8 15491 4167.5 1182.4 411.28
558.57
34 31.09 55.45 70.86
140137 15549 1980.3 1983.2 4058.1
35 36.1 5.3
85.9 18047 8233.2 204.35 3032.9
539.41
36 41.28 53.13 27.9
54349 8965.6 2063.6 945.77 1695.2
37 39.8 93.2
24.7 82683 14523 3555.6
1025.8 2505.5
38 36.4 18.2
24.4 18038 4066.1 639.21 885.99
413.35
39 40.6 79.16 9.97
22304 7091 2543.9 380.58 563.76
40 39.26 68.27 49.03
134719 15234 2542.9 2119.4 3346
41 43.6 58.8
37 65440 9771.6 2041.7
1547.1 1570.3
42 38.2 34
44.9 64215 10101 1364.8
2012.6 1442
43 43.8 16.3
5.4 4199.9 1996.3 575.36 234.26
100.87
44 44.02 82.78 13.64
43630 10486 3277.1 619.66 1224.3
45 39.6 113.17 113.38
417007 33333 4758.7 3786.1 9028.4
46 47.9 95.4
74.8 302758 31069 4292.7 3426.8
7408.8
47 49.4 68.8
41 134357 14481 3692.6
1848.2 2940.6
48 41.8 25.5
18.8 26067 5728.8 1340.7 1108.1
595.44
49 47.05 43.5 39.32
83281 11734 2565 1853.9
1901.2
50 43.5 92.7
10.1 44383 12802 4416.9
529.23 937.23
51 54.07 89.08 81.2
373183 31437 4057.1 4453.2 7762
52 62.8 53.7
55.7 161645 15415 2597.3 2644.7
2744.7
53 54 84
19.4 106116 16168 5031.3 1111.7
1724.8
54 53 45.7
8.1 20557 6417.7 2094.3
436.29 377.05
55 52.1 102.33 65.63
349367 29094 4956.6 3115.3 6368.6
56 49.6 96.9
68.6 339606 26409 4286.6 3274
5975.2
57 57.5 70.4
73.2 290231 22259 3685.7 3425.2
4172.2
58 56.1 93.6
100.81 415969 40711 6393.2 4677.6 7528.2
59 56.5 89.4
26 120585 19195 5627.3
1446.4 1902.9
60 56.2 9.1
11.8 6153.6 3049.5 511.09 777.65
100.37
61 53.92 39.19 62.2
141252 16954 2238.6 3760.6 2159
62 60.5 61.6
24 74893 12036 3602.1
1697.7 1466.1
63 58.7 82.7
21.8 105463 17706 5106.8 1354.7
1842.4
64 74.1 56
94.3 352019 27633 4158.5 5852.8
4726.3
65 67.7 60.7
25.8 78513 13935 3365.2
1556 1262.4
66 65 35.8
39.2 92111 10761 2151.1
2661.6 1339.4
67 71.6 12.9
22.4 16848 5674.3 936.26 1268.8
271.81
68 60.85 76.91 63.22
306231 33897 5536.6 4940.5 5254.9
69 69.5 77.6
82.8 442687 37553 5536.4 5428.7
6415
70 70.7 44.7
49 126465 16244 3204.7
3260.7 1995.7
71 77.2 37.8
86 228626 24201 2926.2
6384.9 3361.5
72 65.2 3.8
61.8 21228 12526 288.25
4866.9 291.06
73 78.22 49.99 39.08
166363 20794 3877.6 2840.2 2004.8
74 79.3 66.5
64.5 327944 25728 4483.8 5493
3984.8
75 78.6 88.1
100.23 654582 38543 6396 8423.6
8230.8
76 73.5 5.4
86.7 49546 18389 504.46
6946.3 637.73
77 68.88 76.6 44.05
304937 24676 6480.9 3178.1 3180.4
78 74.3 12.7
21.5 19682 5756.9 911.55 1654.7
223.49
79 74.35 36.44 107.36
335789 27767 2853.4 8284.9 3588.1
80 82.1 56.1
95.8 471415 31064 4674.2 7870.3
5019.1
81 80.2 4
21.5 6521.9 4709.4 296.63 1704.6
85.1
82 84.1 50.8
47.3 241456 24941 5023.6 4167.6
2951.4
83 92 77.7
113.96 626640 51822 6242.7 9204.6 8018.4
84 97 76.3
48.1 367451 28961 6334.3 4993.8
4092.1
85 88 3
17 4872.8 3708.9 277.81
1571.3 50.8
86 84.7 62.2
33.9 217114 22154 6334.1 2728.5
2735.7
87 88.7 85.1
42.4 368686 31560 9472.6 3650.2
3874
88 66.6 25
87.9 203989 22467 2794.6 7459.9
2283.7
89 83.6 67.8
13.5 74769 14932 5698
1286.3 795.73
90 92.41 40.21 86.46
351389 32123 3351.4 7702.8 4021
91 102.07 50.62 72.26
339999 30623 4966.3 6730.9 3460.6
92 101.88 9.32 1.83
1911.2 1910.4 810.19 163.29 20.47
93 94.7 47.15 101.58
485579 36252 5281.7 9182.2 4518.5
94 96.4 22.3
22.3 42011 10556 2202
2168 602.14
95 99.31 42.94 28.71
115529 18530 4541.4 2273.5 1087.7
96 90.7 1
61.2 5784.2 11159 100.61 6301.5
55.2
97 82.8 111.18 87.5
870213 49799 11165 8535.3 7745.7
98 91.5 95.6
67.9 529609 39637 10288
6320.7 5567.4
99 104.8 24.7 68.63
216657 20685 3087.6 7246.5 2140.8
100 95.2 47.1 88.7
402881 35965 4814.3 9094.9 3871.1
Mittelwert Varianz
Standard AW
1 50.4
847.6 29.1
2 54.9
852.9 29.2
3 51.8
815.7 28.6
4 151701.9
29554781600 171915
5 16405.6
128724150.5 11345.7
6 2757.7
5394369.8 2322.6
7 2698.5
6446189.3 2538.9
8 2861
5182826.4 2276.6
Anmerkung: Beispiel, daß die großen Unterschiede zwischen den Zahlen für die Korrelationen invariant sind hier.
[Interne Quellen: 30.10.2002 fuer Batchbetrieb Kor
fuer Quaderversuch
Daten von C:\OMI\NUMERIK\MATRIX\URDAT\BASIS\QF50\QF50v15
Dateiname = C:\OMI\NUMERIK\MATRIX\URDAT\BASIS\QF50v15\KOR\QF50v15.DAN
Korrelation in C:\OMI\NUMERIK\MATRIX\URDAT\BASIS\QF50v15\KOR\.d08
Auswertung vom 11/05/02 16:41:26]
Standard-Matrixanalyse
der Korrelationsmatrix dieses Beispiels F50.15
| Abstract/ Zusammenfassung Analsye Korrelationsmatrix
der Basisdaten
Die Matrix ist wie die ganze Serie positiv definit, d.h. es kommen keine bösartigen Entgleisungen durch Kollinearitäts- und rundungsbedingte Instabilitäten vor. |
Samp _Ord_
MD_
NumS_
Condition_
Determinant_
HaInRatio_
R_OutIn_
K_Norm_
C_Norm
100 8 0 --
4567.7 1.19D-7 8.99D-13
2270.9 1D-3(1) .056(1)
|
|
********** Summary
of standard correlation matrix analysis ***********
File = QF50v15.d08 N-order= 8 N-sample=
100 Rank= 8 Missing data = 0
Positiv Definit=Cholesky successful________= Yes with 0 negat.
eigenvalue/s
HEVA: Highest eigenvalue abs.value_________=
5.0494490113276507
LEVA: Lowest eigenvalue absolute value_____=
1.1054739134424493D-3
CON: Condition number HEVA/LEVA___________~=
4567.6781242206342
DET: Determinant original matrix (OMIKRON)_=
1.191587531929124D-7
DET: Determinant (CHOLESKY-Diagonal^2)_____=
1.191587531929124D-7
DET: Determinant (PESO-CHOLESKY)___________=
1.191587531929124D-7
DET: Determinant (product eigenvalues)_____=
1.1915875319291179D-7
DET: Determ.abs.val.(PESO prod.red.norms)__=
1.1915875319291239D-7
HAC: HADAMARD condition number_____________=
7.7110164779836343D-10
HCN: Heuristic condition |DET|CON__________=
2.6087379616584522D-11
D_I: Determinant Inverse absolute value____= 8392166
HDA: HADAMARD Inequality absolute value___<=
9.3286357763311021D+18
HIR: HADAMARD RATIO: D_I / HDA ____________=
8.9961339358334676D-13
Highest inverse positive diagonal value____=
318.880585599
thus multiple r( 5.rest)_________________=
.998430784
and 5 multiple r > .99
There are no negative inverse diagonal values.
Maximum range (upp-low) multip-r( 4.rest)_=
.242
LES: Numerical stability analysis:
Ratio maximum range output / input _______=
2270.8950811337616
PESO-Analysis correlation least Ratio RN/ON=
8.72D-4 (<-> Angle = .05 )
Number of Ratios correlation RN/ON < .01__ =
1
PESO-Analysis Cholesky least Ratio RN/ON__ =
.056 (<-> Angle = 3.21 )
Number of Ratios Cholesky RN/ON < .1 _____ =
1
Ncor
L1-Norm L2-Norm Max Min
m|c| M|c| N_comp s-S
S-S
64 39.2 5.4
1 -.099 .556 .26
378 .296 .23
class boundaries and distribution of the correlation coefficients
-1 -.8 -.6 -.4 -.2 0
.2 .4 .6 .8 1
0 0 0
0 2 8 2
14 20 18
Original data with 17, input read with 17, computet
with 19,
and showed with 3 digit accuracy
(for control here the analysed original matrix):
Läng Breit Höhe Volum Ober
L*B L*H B*H
1 -.099 .102 .545 .557
.606 .706 .041
-.099 1 .153 .482 .542
.605 .054 .727
.102 .153 1 .628
.668 .242 .695 .711
.545 .482 .628 1
.96 .84 .822 .752
.557 .542 .668 .96 1
.842 .831 .795
.606 .605 .242 .84 .842
1 .579 .529
.706 .054 .695 .822 .831 .579
1 .463
.041 .727 .711 .752 .795 .529
.463 1
i.Eigenvalue Cholesky i.Eigenvalue
Cholesky i.Eigenvalue Cholesky
1. 5.04945 1
2. 1.5495 .9951
3. 1.09273 .9812
4. .13847 .4113
5. .08269 .1503
6. .07043 .3069
7. .01563 .2559
8. 1.11D-3 .0728
Cholesky decomposition successful, thus the matrix is (semi)
positive definit.
Eigenwerte in Prozentanteilen von der Spur = 8
1 .6312 2 .1937 3 .1366
4 .0173 5 .0103 6 8.8D-3
7 2D-3 8 1D-4 (mit 100 multiplizieren)
[Interne Quelle: analysed: 11/12/02 12:15:26
PRG version 30.10.2002 MABAT9q.BAS
File = C:\OMI\NUMERIK\MATRIX\SMA\QF\QF50v15\QF50v15.SMA
with data from C:\OMI\NUMERIK\MATRIX\SMA\QF\QF50v15\QF50v15.d08]
Die drei Hauptfaktoren und Rückrechnung der Reproduktionsmatrix dieses Beispiels
Matrix der 8 Faktoren nach VEK * EIW^1/2:
.551 -.744 -.296 -.22 .031 -.064 .034 -3D-3
.542 .714 -.404 -.082 -.154 1D-3 .041 -5D-3
.684 .09 .706 .038 -.027 -.148
.04 -4D-3
.974 -.043 -.025 .125 .113 .136 .055
-.01
.996 -6D-3 -.016 -.01 -.018 -.013 -.086 -.019
.838 -.042 -.497 .189 .029 -.115 -.011
.014
.834 -.45 .25 .012 -.163 .108
-.011 .014
.804 .52 .183 -.174 .13
.028 -.022 .015
Transponierte der 8 Faktoren:
.551 .542 .684 .974 .996 .838
.834 .804
-.744 .714 .09 -.043 -6D-3 -.042 -.45
.52
-.296 -.404 .706 -.025 -.016 -.497 .25
.183
-.22 -.082 .038 .125 -.01 .189
.012 -.174
.031 -.154 -.027 .113 -.018 .029 -.163
.13
-.064 1D-3 -.148 .136 -.013 -.115 .108
.028
.034 .041 .04 .055 -.086 -.011 -.011
-.022
-3D-3 -5D-3 -4D-3 -.01 -.019 .014 .014
.015
Reproduktionsmatrix
aus allen 8 Faktoren:
1 -.099 .102 .545 .557
.606 .706 .041
-.099 1 .153 .482 .542
.605 .054 .727
.102 .153 1 .628
.668 .242 .695 .711
.545 .482 .628 1
.96 .84 .822 .752
.557 .542 .668 .96 1
.842 .831 .795
.606 .605 .242 .84 .842
1 .579 .529
.706 .054 .695 .822 .831 .579
1 .463
.041 .727 .711 .752 .795 .529
.463 1
Residualanalyse Original-Korrelationen und aus 8 Faktoren reproduzierte:
0 0 0
0 0 0
0 0
0 0 0
0 0 0
0 0
0 0 0
0 0 0
0 0
0 0 0
0 0 0
0 0
0 0 0
0 0 0
0 0
0 0 0
0 0 0
0 0
0 0 0
0 0 0
0 0
0 0 0
0 0 0
0 0
Mittelwert der absoluten Abweichungen .. = 8.3795910680683865D-18
Standardabweichung der abs. Abweichungen = 5.223809979403365D-18
Maximaler absoluter Abweichungswert .... = 2.7321894746634712D-17
Reproduktionsmatrix aus 3 Faktoren:
.945 -.113 .101 .576 .558
.639 .721 1D-3
-.113 .968 .15 .508 .542
.625 .03 .734
.101 .15 .974 .644 .669
.219 .707 .727
.576 .508 .644 .95 .97
.83 .825 .756
.558 .542 .669 .97 .992
.842 .829 .795
.639 .625 .219 .83 .842
.95
.594 .561
.721 .03 .707 .825 .829
.594 .961 .483
1D-3 .734 .727 .756 .795 .561
.483 .951
Residualanalyse Original-Korrelationen und aus 3 Faktoren reproduzierte:
.055 .015 2D-3 .031 0
.034 .015 .04
.015 .032 2D-3 .025 0
.021 .024 7D-3
2D-3 2D-3 .026 .016 1D-3 .023
.012 .015
.031 .025 .016 .05 .01
.011 3D-3 5D-3
0 0 1D-3
.01 8D-3 0 2D-3 1D-3
.034 .021 .023 .011 0
.05 .015 .032
.015 .024 .012 3D-3 2D-3 .015
.039 .02
.04 7D-3 .015 5D-3 1D-3
.032 .02 .049
Mittelwert der absoluten Abweichungen .. = .016657843237610374
Standardabweichung der abs. Abweichungen = .01450098447537691
Maximaler absoluter Abweichungswert .... = .054816313580873143
[Interne Quellen: Faktoren in C:\OMI\NUMERIK\MATRIX\SMA\QF\QF50v15\FAK\QF50v15.PFA
als 1-zeiliger Spaltenvektor in C:\OMI\NUMERIK\MATRIX\SMA\QF\QF50v15\FAK\QF50v15.FAK
Vektordaten von C:\OMI\NUMERIK\MATRIX\SMA\QF\QF50v15\QF50v15.VEK
Negative Eigenwerte wurden 0 gesetzt:
Eigenwerte von C:\OMI\NUMERIK\MATRIX\SMA\QF\QF50v15\QF50v15.EDI]
Reproduktionen der Originalkorrelationsmatrix aus den drei konstituierenden Faktoren des Quaders dieser 30 Fehlerversuche F50.1-30
Bemerkung: Kleine negative Eigenwerte - meist eine Folge der Kollinearität im Zusammenhang mit Rundungsfehlern -kamen in der 50%-Fehlrspannweitenserie nicht vor, mußten hier also auch nicht 0 gesetzt werden, um die einfache Haupkomponentenmethode nach Cooley & Lohnes durchzuführen. . Zur Methode der Residualbestimmung
Reproduktionsmatrix F50v01 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.952 -.103 .117 .575 .562
.657 .711 .028
-.103 .954 .128 .516 .552
.613 .055 .734
.117 .128 .964 .627 .649
.175 .721 .696
.575 .516 .627 .94 .963
.813 .821 .764
.562 .552 .649 .963 .987
.829 .829 .802
.657 .613 .175 .813 .829 .951
.582 .55
.711 .055 .721 .821 .829 .582
.956
.51
.028 .734 .696 .764 .802 .55
.51 .942
Mittelwert der absoluten Abweichungen .. = .018266129429672806
Standardabweichung der abs. Abweichungen = .014812481831675192
Maximaler absoluter Abweichungswert .... = .059876752740494187
Reproduktionsmatrix F50v02 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.944 -.149 .081 .519 .515
.601 .692 -.054
-.149 .956 .134 .513 .519
.633 7D-3 .712
.081 .134 .969 .653 .687
.204 .714 .724
.519 .513 .653 .947 .966
.815 .803 .758
.515 .519 .687 .966 .987
.819 .824 .784
.601 .633 .204 .815 .819 .952
.557 .539
.692 7D-3 .714 .803 .824 .557
.954
.467
-.054 .712 .724 .758 .784 .539
.467 .941
Mittelwert der absoluten Abweichungen .. = .019062043823446434
Standardabweichung der abs. Abweichungen = .01510880635466133
Maximaler absoluter Abweichungswert .... = .058784086736441265
Reproduktionsmatrix F50v03 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.946 -.12 .068 .578 .565
.641 .737 .021
-.12 .956 .079 .511 .524
.616 9D-3 .7
.068 .079 .971 .564 .61
.139 .659 .701
.578 .511 .564 .945 .966
.839 .806 .759
.565 .524 .61 .966 .99
.839 .825 .797
.641 .616 .139 .839 .839 .952
.593 .553
.737 9D-3 .659 .806 .825 .593
.957
.487
.021 .7 .701 .759 .797
.553 .487 .945
Mittelwert der absoluten Abweichungen .. = .018264280013295097
Standardabweichung der abs. Abweichungen = .014529574360115369
Maximaler absoluter Abweichungswert .... = .055473218788091245
Reproduktionsmatrix F50v04 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.942 -.112 .103 .571 .557
.648 .71 .022
-.112 .958 .134 .499 .53
.61 .033 .712
.103 .134 .968 .628 .667
.203 .715 .721
.571 .499 .628 .929 .957
.817 .816 .752
.557 .53 .667 .957 .987
.83 .833 .798
.648 .61 .203 .817 .83
.946
.59 .551
.71 .033 .715 .816 .833
.59 .959 .508
.022 .712 .721 .752 .798 .551
.508 .936
Mittelwert der absoluten Abweichungen .. = .019777491266888438
Standardabweichung der abs. Abweichungen = .016273422214505437
Maximaler absoluter Abweichungswert .... = .070990516799494391
Reproduktionsmatrix F50v05 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.952 -.119 .086 .583 .535
.637 .714 .029
-.119 .952 .081 .484 .523
.619 .013 .694
.086 .081 .965 .6
.653 .207 .696 .706
.583 .484 .6 .946
.964 .848 .822 .763
.535 .523 .653 .964 .989
.845 .825 .822
.637 .619 .207 .848 .845 .954
.608 .593
.714 .013 .696 .822 .825 .608
.955
.515
.029 .694 .706 .763 .822 .593
.515 .946
Mittelwert der absoluten Abweichungen .. = .01971060099341887
Standardabweichung der abs. Abweichungen = .014024286802739069
Maximaler absoluter Abweichungswert .... = .054321124754758419
Reproduktionsmatrix F50v06 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.943 -.145 .112 .543 .522
.622 .712 -6D-3
-.145 .947 .089 .502 .517
.611 .041 .701
.112 .089 .967 .624 .677
.212 .713 .708
.543 .502 .624 .942 .965
.836 .825 .769
.522 .517 .677 .965 .992
.834 .846 .814
.622 .611 .212 .836 .834 .953
.621 .569
.712 .041 .713 .825 .846 .621
.959
.518
-6D-3 .701 .708 .769 .814 .569
.518 .949
Mittelwert der absoluten Abweichungen .. = .020162537442783721
Standardabweichung der abs. Abweichungen = .01478750672756923
Maximaler absoluter Abweichungswert .... = .058208279204252963
Reproduktionsmatrix F50v07 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.952 -.098 .085 .573 .55
.657 .705 .019
-.098 .957 .102 .489 .517
.61 .056 .7
.085 .102 .971 .633 .671
.215 .711 .719
.573 .489 .633 .946 .966
.831 .845 .765
.55 .517 .671 .966 .99
.836 .855 .808
.657 .61 .215 .831 .836
.945
.631 .568
.705 .056 .711 .845 .855 .631
.962
.533
.019 .7 .719 .765 .808
.568 .533 .947
Mittelwert der absoluten Abweichungen .. = .017929284198597656
Standardabweichung der abs. Abweichungen = .014148670944061728
Maximaler absoluter Abweichungswert .... = .054750612246710377
Reproduktionsmatrix F50v08 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.948 -.146 .113 .56 .559
.642 .723 .022
-.146 .955 .096 .473 .52
.598 -.01 .709
.113 .096 .97 .64
.648 .187 .701 .709
.56 .473 .64 .937
.965 .81 .811 .772
.559 .52 .648 .965 .996
.844 .817 .809
.642 .598 .187 .81 .844
.956
.582 .563
.723 -.01 .701 .811 .817 .582
.948
.49
.022 .709 .709 .772 .809 .563
.49 .952
Mittelwert der absoluten Abweichungen .. = .018356416993688478
Standardabweichung der abs. Abweichungen = .015076580761908134
Maximaler absoluter Abweichungswert .... = .06309451330093843
Reproduktionsmatrix F50v09 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.952 -.122 .102 .592 .583
.645 .735 -1D-3
-.122 .952 .152 .502 .51
.614 .018 .727
.102 .152 .974 .622 .666
.189 .693 .726
.592 .502 .622 .948 .968
.829 .817 .74
.583 .51 .666 .968 .99
.828 .839 .773
.645 .614 .189 .829 .828 .955
.58 .538
.735 .018 .693 .817 .839 .58
.96 .467
-1D-3 .727 .726 .74 .773 .538
.467 .946
Mittelwert der absoluten Abweichungen .. = .017769778788086566
Standardabweichung der abs. Abweichungen = .013978429533744673
Maximaler absoluter Abweichungswert .... = .054101415642454927
Reproduktionsmatrix F50v10 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.952 -.105 .084 .575 .569
.672 .716 2D-3
-.105 .961 .16 .512 .53
.598 .053 .735
.084 .16 .969 .641 .666
.222 .698 .721
.575 .512 .641 .952 .971
.837 .833 .756
.569 .53 .666 .971 .99
.846 .845 .784
.672 .598 .222 .837 .846 .954
.628 .55
.716 .053 .698 .833 .845 .628
.958
.492
2D-3 .735 .721 .756 .784 .55
.492 .943
Mittelwert der absoluten Abweichungen .. = .017653707643867227
Standardabweichung der abs. Abweichungen = .013402023446211292
Maximaler absoluter Abweichungswert .... = .056871235096027572
Reproduktionsmatrix F50v11 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.957 -.095 .057 .545 .549
.655 .69 1D-3
-.095 .957 .092 .516 .523
.617 .015 .719
.057 .092 .959 .614 .646
.187 .694 .691
.545 .516 .614 .943 .966
.826 .799 .774
.549 .523 .646 .966 .991
.836 .824 .799
.655 .617 .187 .826 .836 .945
.584 .568
.69 .015 .694 .799 .824
.584 .944 .484
1D-3 .719 .691 .774 .799 .568
.484 .947
Mittelwert der absoluten Abweichungen .. = .019822242986755235
Standardabweichung der abs. Abweichungen = .014603531409652038
Maximaler absoluter Abweichungswert .... = .057363074471897763
Reproduktionsmatrix F50v12 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.958 -.05 .113 .61 .578
.691 .727 .055
-.05 .963 .107 .511 .549
.619 .031 .721
.113 .107 .966 .618 .666
.188 .712 .7
.61 .511 .618 .942 .967
.825 .819 .756
.578 .549 .666 .967 .996
.828 .828 .813
.691 .619 .188 .825 .828 .945
.59 .552
.727 .031 .712 .819 .828 .59
.964 .494
.055 .721 .7 .756 .813
.552 .494 .942
Mittelwert der absoluten Abweichungen .. = .016772085001164801
Standardabweichung der abs. Abweichungen = .015000918094713479
Maximaler absoluter Abweichungswert .... = .057819799236703113
Reproduktionsmatrix F50v13 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.95 -.133 .096 .554 .554
.647 .716 -4D-3
-.133 .959 .121 .527 .539
.598 .051 .749
.096 .121 .966 .59 .639
.158 .699 .678
.554 .527 .59 .927 .956
.818 .805 .752
.554 .539 .639 .956 .989
.826 .837 .79
.647 .598 .158 .818 .826 .945
.595 .541
.716 .051 .699 .805 .837 .595
.955
.487
-4D-3 .749 .678 .752 .79 .541
.487 .944
Mittelwert der absoluten Abweichungen .. = .018405416800523231
Standardabweichung der abs. Abweichungen = .01522925150748144
Maximaler absoluter Abweichungswert .... = .073392059275795114
Reproduktionsmatrix F50v14 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.944 -.128 .129 .585 .556
.632 .734 .029
-.128 .957 .126 .488 .54
.619 .014 .718
.129 .126 .971 .636 .665
.181 .706 .717
.585 .488 .636 .942 .966
.811 .82 .757
.556 .54 .665 .966 .994
.827 .82 .81
.632 .619 .181 .811 .827 .952
.569 .552
.734 .014 .706 .82 .82
.569 .957 .49
.029 .718 .717 .757 .81
.552 .49 .945
Mittelwert der absoluten Abweichungen .. = .018274534457173636
Standardabweichung der abs. Abweichungen = .015473858168063075
Maximaler absoluter Abweichungswert .... = .057744965229349658
Reproduktionsmatrix F50v15 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
Vollständiges Beispiel siehe bitte oben
.945 -.113 .101 .576 .558
.639 .721 1D-3
-.113 .968 .15 .508 .542
.625 .03 .734
.101 .15 .974 .644 .669
.219 .707 .727
.576 .508 .644 .95 .97
.83 .825 .756
.558 .542 .669 .97 .992
.842 .829 .795
.639 .625 .219 .83 .842
.95
.594 .561
.721 .03 .707 .825 .829
.594 .961 .483
1D-3 .734 .727 .756 .795 .561
.483 .951
Mittelwert der absoluten Abweichungen .. = .016657843237610374
Standardabweichung der abs. Abweichungen = .01450098447537691
Maximaler absoluter Abweichungswert .... = .054816313580873143
Reproduktionsmatrix F50v16 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.946 -.113 .075 .584 .553
.658 .708 2D-3
-.113 .964 .141 .489 .532
.604 .017 .706
.075 .141 .969 .623 .659
.236 .696 .741
.584 .489 .623 .943 .963
.848 .821 .743
.553 .532 .659 .963 .988
.856 .823 .794
.658 .604 .236 .848 .856 .955
.616 .559
.708 .017 .696 .821 .823 .616
.955
.494
2D-3 .706 .741 .743 .794 .559
.494 .945
Mittelwert der absoluten Abweichungen .. = .018029873356042811
Standardabweichung der abs. Abweichungen = .014293545310381074
Maximaler absoluter Abweichungswert .... = .057271669296253455
Reproduktionsmatrix F50v17 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.944 -.115 .109 .589 .574
.643 .721 .014
-.115 .952 .112 .494 .523
.617 -2D-3 .716
.109 .112 .97 .61
.649 .187 .703 .706
.589 .494 .61 .939 .965
.829 .803 .743
.574 .523 .649 .965 .993
.84 .818 .788
.643 .617 .187 .829 .84
.953
.567 .554
.721 -2D-3 .703 .803 .818 .567
.952
.47
.014 .716 .706 .743 .788 .554
.47 .943
Mittelwert der absoluten Abweichungen .. = .018904339158656048
Standardabweichung der abs. Abweichungen = .015251983470225178
Maximaler absoluter Abweichungswert .... = .060681885108944258
Reproduktionsmatrix F50v18 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.962 -.1 .127 .569 .552
.625 .737 .046
-.1 .955 .129 .528 .544
.651 .041 .703
.127 .129 .965 .64 .675
.195 .707 .733
.569 .528 .64 .947 .964
.817 .813 .786
.552 .544 .675 .964 .983
.817 .825 .82
.625 .651 .195 .817 .817 .949
.571 .57
.737 .041 .707 .813 .825 .571
.957
.519
.046 .703 .733 .786 .82
.57 .519 .949
Mittelwert der absoluten Abweichungen .. = .017848482399493869
Standardabweichung der abs. Abweichungen = .013486782171006848
Maximaler absoluter Abweichungswert .... = .053208626764459002
Reproduktionsmatrix F50v19 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.954 -.118 .085 .572 .56
.659 .713 .047
-.118 .958 .136 .504 .523
.598 7D-3 .743
.085 .136 .967 .625 .664
.178 .7 .695
.572 .504 .625 .943 .966
.816 .807 .782
.56 .523 .664 .966 .991
.821 .825 .818
.659 .598 .178 .816 .821 .945
.571 .581
.713 7D-3 .7 .807 .825
.571 .955 .492
.047 .743 .695 .782 .818 .581
.492 .95
Mittelwert der absoluten Abweichungen .. = .018690886926366067
Standardabweichung der abs. Abweichungen = .014219339514942411
Maximaler absoluter Abweichungswert .... = .056589302927650869
Reproduktionsmatrix F50v20 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.952 -.104 .087 .568 .571
.643 .736 .02
-.104 .956 .082 .516 .505
.633 .039 .692
.087 .082 .969 .592 .651
.194 .678 .713
.568 .516 .592 .939 .965
.848 .817 .765
.571 .505 .651 .965 .994
.846 .856 .797
.643 .633 .194 .848 .846 .962
.628 .576
.736 .039 .678 .817 .856 .628
.962
.509
.02 .692 .713 .765 .797
.576 .509 .946
Mittelwert der absoluten Abweichungen .. = .01807757792996782
Standardabweichung der abs. Abweichungen = .014005810278950757
Maximaler absoluter Abweichungswert .... = .060982690495497215
Reproduktionsmatrix F50v21 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.95 -.128 .096 .566 .558
.625 .73 .045
-.128 .96 .135 .534 .557
.626 .077 .732
.096 .135 .966 .605 .631
.162 .69 .707
.566 .534 .605 .951 .972
.826 .834 .789
.558 .557 .631 .972 .994
.837 .846 .82
.625 .626 .162 .826 .837 .946
.604 .578
.73 .077 .69 .834 .846
.604 .965 .542
.045 .732 .707 .789 .82
.578 .542 .949
Mittelwert der absoluten Abweichungen .. = .017566794159826596
Standardabweichung der abs. Abweichungen = .014938137620890068
Maximaler absoluter Abweichungswert .... = .054183877622267525
Reproduktionsmatrix F50v22 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.96 -.132 .109 .581 .593
.653 .748 .049
-.132 .959 .108 .495 .487
.599 .01 .724
.109 .108 .963 .613 .652
.153 .685 .699
.581 .495 .613 .941 .964
.811 .816 .778
.593 .487 .652 .964 .989
.814 .849 .799
.653 .599 .153 .811 .814 .949
.585 .561
.748 .01 .685 .816 .849
.585 .961 .5
.049 .724 .699 .778 .799 .561
.5 .952
Mittelwert der absoluten Abweichungen .. = .017784564137978517
Standardabweichung der abs. Abweichungen = .013387685589163975
Maximaler absoluter Abweichungswert .... = .059053181639259361
Reproduktionsmatrix F50v23 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.949 -.144 .037 .568 .534
.649 .694 .017
-.144 .957 .136 .482 .518
.589 -3D-3 .718
.037 .136 .966 .608 .645
.196 .679 .714
.568 .482 .608 .95 .966
.842 .814 .769
.534 .518 .645 .966 .986
.844 .816 .814
.649 .589 .196 .842 .844 .954
.594 .585
.694 -3D-3 .679 .814 .816 .594
.947
.499
.017 .718 .714 .769 .814 .585
.499 .944
Mittelwert der absoluten Abweichungen .. = .0187676383385313
Standardabweichung der abs. Abweichungen = .013869291164396824
Maximaler absoluter Abweichungswert .... = .056050450198512611
Reproduktionsmatrix F50v24 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.943 -.116 .08 .547 .522
.628 .699 -.02
-.116 .957 .139 .517 .552
.631 .066 .739
.08 .139 .97 .638
.672 .189 .712 .702
.547 .517 .638 .945 .967
.816 .834 .758
.522 .552 .672 .967 .992
.825 .841 .803
.628 .631 .189 .816 .825 .951
.596 .555
.699 .066 .712 .834 .841 .596
.963
.498
-.02 .739 .702 .758 .803 .555
.498 .943
Mittelwert der absoluten Abweichungen .. = .018087183748891812
Standardabweichung der abs. Abweichungen = .015056157270502826
Maximaler absoluter Abweichungswert .... = .056809080207424038
Reproduktionsmatrix F50v25 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.955 -.097 .087 .605 .59
.667 .738 .056
-.097 .96 .14 .504 .542
.613 .026 .725
.087 .14 .971 .601 .632
.169 .676 .725
.605 .504 .601 .942 .967
.829 .816 .765
.59 .542 .632 .967 .995
.845 .825 .809
.667 .613 .169 .829 .845 .955
.589 .564
.738 .026 .676 .816 .825 .589
.955
.503
.056 .725 .725 .765 .809 .564
.503 .955
Mittelwert der absoluten Abweichungen .. = .016724948771071383
Standardabweichung der abs. Abweichungen = .01402153444938972
Maximaler absoluter Abweichungswert .... = .057864850311864051
Reproduktionsmatrix F50v26 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.948 -.112 .094 .565 .552
.654 .721 .022
-.112 .96 .149 .529 .554
.61 .047 .749
.094 .149 .966 .615 .655
.18 .698 .706
.565 .529 .615 .937 .962
.821 .81 .77
.552 .554 .655 .962 .99
.829 .828 .812
.654 .61 .18 .821 .829
.949
.592 .56
.721 .047 .698 .81 .828
.592 .96 .497
.022 .749 .706 .77 .812
.56 .497 .955
Mittelwert der absoluten Abweichungen .. = .017919051013848818
Standardabweichung der abs. Abweichungen = .014723542088411136
Maximaler absoluter Abweichungswert .... = .063361453013835167
Reproduktionsmatrix F50v27 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.952 -.115 .06 .53 .538
.627 .688 8D-3
-.115 .952 .166 .54 .554
.634 .044 .735
.06 .166 .97 .621
.659 .214 .707 .724
.53 .54 .621 .926
.958 .828 .791 .777
.538 .554 .659 .958 .991
.846 .822 .811
.627 .634 .214 .828 .846 .952
.579 .589
.688 .044 .707 .791 .822 .579
.953
.508
8D-3 .735 .724 .777 .811 .589
.508 .947
Mittelwert der absoluten Abweichungen .. = .020196289800343188
Standardabweichung der abs. Abweichungen = .014905195037714982
Maximaler absoluter Abweichungswert .... = .073961462775978028
Reproduktionsmatrix F50v28 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.954 -.12 .074 .579 .565
.66 .711 .024
-.12 .954 .123 .484 .518
.6 7D-3 .696
.074 .123 .973 .627 .651
.221 .694 .737
.579 .484 .627 .95 .971
.845 .82 .768
.565 .518 .651 .971 .994
.86 .827 .805
.66 .6 .221 .845
.86 .959 .61 .575
.711 7D-3 .694 .82 .827
.61 .951 .509
.024 .696 .737 .768 .805 .575
.509 .948
Mittelwert der absoluten Abweichungen .. = .017943086137502446
Standardabweichung der abs. Abweichungen = .013552036031526695
Maximaler absoluter Abweichungswert .... = .051835586222441811
Reproduktionsmatrix F50v29 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.956 -.101 .15 .597 .604
.662 .761 .057
-.101 .957 .117 .496 .498
.612 8D-3 .695
.15 .117 .969 .648 .682
.213 .695 .734
.597 .496 .648 .947 .97
.823 .817 .772
.604 .498 .682 .97 .995
.831 .843 .797
.662 .612 .213 .823 .831 .95
.6 .554
.761 8D-3 .695 .817 .843 .6
.958 .493
.057 .695 .734 .772 .797 .554
.493 .947
Mittelwert der absoluten Abweichungen .. = .017070063019682471
Standardabweichung der abs. Abweichungen = .013618754462318341
Maximaler absoluter Abweichungswert .... = .053413190447011868
Reproduktionsmatrix F50v30 aus 3 Faktoren und
Vergleich mit der Original Korrelationsmatrix F50
.953 -.144 .079 .557 .549
.633 .722 .014
-.144 .958 .112 .494 .53
.607 .019 .711
.079 .112 .966 .622 .633
.165 .684 .714
.557 .494 .622 .948 .968
.815 .823 .777
.549 .53 .633 .968 .989
.836 .826 .807
.633 .607 .165 .815 .836 .949
.588 .558
.722 .019 .684 .823 .826 .588
.955
.507
.014 .711 .714 .777 .807 .558
.507 .949
Mittelwert der absoluten Abweichungen .. = .018133132376307597
Standardabweichung der abs. Abweichungen = .01386434611354488
Maximaler absoluter Abweichungswert .... = .052498240463983674
Statistik der Eigenwerte im 50% Fehlerspannweiten Versuch
| Die Eigenwerte sind die 'Gene' und 'Herzstück' einer Korrelationsmatrix. Kennt man die Eigenwerte, weiß man alles Wesentliche über die Gesetz- und Regelhaftigkeiten der Korrelationsmatrix und wie viele Fast- Kollinearitäten, d.h. fast-funktionale Abhängigkeiten sie enthält, um so mehr, je mehr Eigenwerte 'nahe' 0 sich in der Korrelations- Matrix finden. Jede ForscherIn sollte daher bestrebt sein, Eigenwerte 'nahe' 0 aufzuspüren, Theorien, Modelle und Hypothesenprüfungen entwickeln, um diese Gesetzmäßigkeiten zu erklären. Nur FaktorenaanlytikerInnen interessieren sich anscheinend nicht nur nicht dafür, sie definieren und beschließen einfach wie numerologische Metaphysiker durch ein narzißtisches Orakel, daß so und so viele Eigenwerte per definitionem 0 gesetzt werden und dann meist auch "weggeworfen" werden, ein ganz absonderliches wissenschaftliches Gebaren. Dabei verkennen sie, daß die Korrelationmatrix zu den den zentriert-normierten Rohdaten in isometrischer Relation steht (Hain 1994, Kap. 6, S. 20, Satz 3.4), d.h. eine gewaltsame 0-Setzung ist einer Datenverfälschung äquivalent. Gewaltsam heißt hier, daß die Matrix nicht in ihrer empirischen Erscheinung von vorneherein Eigenwerte 'nahe' 0 enthält. |
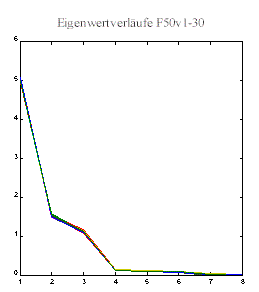 |
Der Graph aller
30 Eigenwertverläufe
bei der Fehlerspannweite von 50% (WW + - 25%) demonstriert eindrucksvoll, wie nahe alle 30 Eigenwertverläufe beieinanderliegen. Man kann die 30 Linien gar nicht richtig erkennen, so gering ist die Streuung. Dies zeigt auch klar, daß die üblichen Phantasien, Rationalisierungen und "Argumente" der FaktorenanalytikerInnen, wie sich zufällige Fehler in Eigenwertverläufen graphisch darstellen, wenigstens für den Typ Quader 8V3u5a falsch sind. Die beiden großen Flächen unter den beiden Knicken (2, 3) repräsentieren ganz unzweideutig die drei unabhängigen Variablen und die restlichen fünf (4,5,6,7,8) stehen für die linear abhängigen Variablen. |
Mittelwert,
Sigma, Max und Min der Eigenwerte der Serie:
Mit 5.0180 1.5280 1.1175 0.1317 0.1065
0.0752 0.0163 0.0068
Sig 0.0365 0.0256 0.0199 0.0092 0.0077
0.0072 0.0049 0.0032
Max 5.0740 1.5819 1.1756 0.1535 0.1200
0.0989 0.0313 0.0153
Min 4.9427 1.4718 1.0729 0.1160 0.0827
0.0655 0.0077 0.0011
Eigenwerte der Serie
F50v1-30:
F50v01 5.0258 1.4985 1.1215 0.1232
0.1154 0.0788 0.0216 0.0153
F50v02 4.9427 1.5819 1.1257 0.1426
0.1066 0.0765 0.0155 0.0086
F50v03 4.9496 1.5355 1.1756 0.1338
0.1065 0.0716 0.0213 0.0061
F50v04 5.0107 1.5066 1.1085 0.1457
0.1200 0.0748 0.0263 0.0074
F50v05 5.0230 1.5093 1.1256 0.1367
0.1165 0.0705 0.0142 0.0042
F50v06 5.0268 1.5271 1.0984 0.1535
0.1088 0.0666 0.0125 0.0063
F50v07 5.0686 1.4798 1.1196 0.1322
0.1005 0.0741 0.0138 0.0114
F50v08 4.9841 1.5577 1.1198 0.1348
0.1124 0.0704 0.0190 0.0018
F50v09 4.9984 1.5693 1.1082 0.1252
0.1094 0.0698 0.0124 0.0072
F50v10 5.0661 1.5390 1.0729 0.1189
0.1081 0.0728 0.0168 0.0053
F50v11 4.9696 1.5169 1.1554 0.1238
0.1161 0.0989 0.0138 0.0055
F50v12 5.0588 1.4718 1.1461 0.1273
0.1009 0.0758 0.0159 0.0033
F50v13 4.9605 1.5555 1.1185 0.1301
0.1050 0.0863 0.0313 0.0129
F50v14 5.0084 1.5408 1.1122 0.1388
0.1108 0.0704 0.0131 0.0054
F50v15 5.0494 1.5495 1.0927 0.1385
0.0827 0.0704 0.0156 0.0011
F50v16 5.0315 1.5382 1.0962 0.1407
0.0932 0.0701 0.0185 0.0115
F50v17 4.9735 1.5424 1.1307 0.1361
0.1123 0.0787 0.0197 0.0065
F50v18 5.0599 1.4924 1.1142 0.1237
0.1025 0.0753 0.0208 0.0112
F50v19 5.0126 1.5357 1.1154 0.1302
0.1102 0.0776 0.0119 0.0064
F50v20 5.0381 1.5027 1.1388 0.1316
0.1080 0.0658 0.0116 0.0033
F50v21 5.0740 1.5008 1.1060 0.1420
0.0974 0.0655 0.0077 0.0068
F50v22 4.9992 1.5503 1.1254 0.1203
0.1100 0.0693 0.0153 0.0101
F50v23 4.9844 1.5566 1.1127 0.1181
0.1124 0.0887 0.0189 0.0083
F50v24 5.0215 1.5284 1.1150 0.1388
0.1027 0.0737 0.0116 0.0083
F50v25 5.0429 1.5164 1.1289 0.1241
0.0951 0.0768 0.0113 0.0046
F50v26 5.0283 1.5330 1.1024 0.1315
0.1025 0.0795 0.0163 0.0064
F50v27 5.0267 1.5153 1.1014 0.1433
0.1150 0.0795 0.0145 0.0042
F50v28 5.0462 1.5236 1.1131 0.1312
0.1028 0.0663 0.0126 0.0041
F50v29 5.0730 1.5146 1.0910 0.1160
0.1035 0.0765 0.0219 0.0035
F50v30 4.9851 1.5491 1.1329 0.1190
0.1083 0.0850 0.0138 0.0068
Abweichungen
Eigenwerte der Serie F50v1-30 von den Wahre Wert Eigenwerten
0.0227 0.0276 0.0150 0.0076 0.0149
0.0109 0.0165 0.0153
0.1058 0.0558 0.0108 0.0270 0.0061
0.0086 0.0105 0.0086
0.0989 0.0095 0.0391 0.0182 0.0061
0.0037 0.0162 0.0061
0.0378 0.0195 0.0280 0.0301 0.0195
0.0069 0.0212 0.0074
0.0254 0.0168 0.0109 0.0211 0.0161
0.0026 0.0092 0.0042
0.0216 0.0011 0.0381 0.0379 0.0083
0.0013 0.0074 0.0063
0.0202 0.0462 0.0168 0.0166 0.0000
0.0062 0.0087 0.0114
0.0644 0.0317 0.0167 0.0192 0.0119
0.0025 0.0139 0.0018
0.0500 0.0433 0.0282 0.0096 0.0090
0.0019 0.0073 0.0072
0.0177 0.0129 0.0636 0.0033 0.0077
0.0049 0.0117 0.0053
0.0789 0.0092 0.0190 0.0082 0.0156
0.0310 0.0088 0.0055
0.0103 0.0543 0.0096 0.0117 0.0005
0.0079 0.0109 0.0033
0.0880 0.0294 0.0179 0.0145 0.0045
0.0184 0.0263 0.0129
0.0400 0.0148 0.0242 0.0232 0.0104
0.0025 0.0080 0.0054
0.0010 0.0234 0.0437 0.0229 0.0178
0.0025 0.0105 0.0011
0.0169 0.0122 0.0402 0.0251 0.0073
0.0022 0.0134 0.0115
0.0750 0.0163 0.0057 0.0205 0.0119
0.0109 0.0146 0.0065
0.0114 0.0337 0.0222 0.0081 0.0020
0.0074 0.0158 0.0112
0.0358 0.0096 0.0211 0.0146 0.0097
0.0097 0.0068 0.0064
0.0104 0.0233 0.0024 0.0160 0.0076
0.0021 0.0065 0.0033
0.0255 0.0253 0.0305 0.0264 0.0031
0.0024 0.0026 0.0068
0.0492 0.0243 0.0110 0.0047 0.0096
0.0014 0.0102 0.0101
0.0640 0.0305 0.0237 0.0025 0.0119
0.0208 0.0138 0.0083
0.0269 0.0023 0.0215 0.0232 0.0023
0.0058 0.0065 0.0083
0.0056 0.0097 0.0076 0.0085 0.0054
0.0089 0.0062 0.0046
0.0201 0.0070 0.0340 0.0159 0.0021
0.0116 0.0112 0.0064
0.0217 0.0108 0.0351 0.0277 0.0146
0.0116 0.0094 0.0042
0.0022 0.0024 0.0234 0.0156 0.0023
0.0016 0.0075 0.0041
0.0245 0.0115 0.0455 0.0004 0.0030
0.0086 0.0169 0.0035
0.0633 0.0230 0.0036 0.0034 0.0079
0.0171 0.0087 0.0068
Mittelwert der absoluten Abweichungen .. = .016621303435517882
Standardabweichung der abs. Abweichungen = .016654415320888875
Maximaler absoluter Abweichungswert .... = .1057577637385599
Zusammenfassung/
Abstract Ergebnisse F50v1...30
| Völlig überraschend, entgegen dem gesunden Menschenverstand und vernünftiger Erwartung, ergibt die Versuchsserie mit 50% Fehlerspannweite (WW + - 25%) eine ziemlich gute Reproduzierbarkeit der jeweiligen Ursprungsmatrizen. Dies widerlegt faktorenanalytische Rationalisierungen und Legenden von der Fehlerwirkung, wonach Eigenwerte kleiner 1 sozusagen als "zufallsbedingt" vernachlässigt werden können. In dieser Deutung steckt sogar ein Doppelfehler. Kleine Eigenwerte sind nicht etwa eine Folge zufälliger Einflüsse, sondern Eigenwerte 'nahe' 0 bedeuten eine gesetzesartige Beziehung vom Typ lineare Abhängigkeit, sind also das genaue Gegenteil von "Zufällen" wie diese Versuchsserie auch beweist. Zweitens zeigen die Ergebnisse bislang völlig überraschend, daß zufällige Fehler bis um die Größenordnung von sage und schreibe 100% sich nicht besonders nennenswert auf die Eigenwert- und Faktorenstruktur auswirken [Warnung]. Faktorenanalytische Fehlerphantasien, wie sie auch um den sog. Screetest ranken, sind daher nicht besser als psychoanalytische Phantastereien, sie tragen nur für viele methodisch nicht so Versierte eine schwer angreifbare szientistische Verkleidung [hierzu Glosse und ein bissiger Wissenschaftspolitischer Kommentar]. |
___
Sponsel, Rudolf & Hain, Bernhard (1994). Numerisch instabile Matrizen und Kollinearität in der Psychologie. Diagnose, Relevanz & Utilität, Frequenz, Ätiologie, Therapie. Ill-Conditioned Matrices and Collinearity in Psychology. Deutsch-Englisch. Übersetzt von Agnes Mehl. Kapitel 6 von Dr. Bernhard Hain: Bemerkungen über Korrelationsmatrizen. Erlangen: IEC-Verlag [ISSN-0944-5072 ISBN 3-923389-03-5]
Aktueller Preis: http://ww.iec-verlag.de
___
Man kann die numerische Stabilität einer Korrelationsmatrix erhöhen, wenn man die Diagonalelemente numerisch größer macht. Diese Methode stammt von TIKHONOV, heißt auch Regularisierungs- oder Ridge-Methode. Siehe Sponsel (1994, Kap. 5, S. 08-10) mit Beispielen.
zur methodischen Umgebung dieser Untersuchung:
Standort: Versuch q50.
*
Überblicks- und Verteilerseite zu diesen Versuchen
Vorläufige Hauptergebnisse und Statistik der Eigenwertanalysen
Einführung und Überblick. Kritik der Handhabung der Faktorenanalyse
Was für ein Typ Matrix entsteht durch Faktorenanalysen?
Überblicks- und Verteilerseite: Numerisch instabile Matrizen und Kollinearität in der Psychologie
Glosse zum numerologischen Szientismus in der Psychologie
Bissiger Wissenschaftspolitischer Kommentar gegen den numerologischen Szientismus in der Psychologie
*
Dienstleistungs-Info.
*
Zitierung
Sponsel, Rudolf (DAS). 30 Versuche mit zufällig normalverteilter Fehlerspannweite von 50% bezüglich der definierten wahren Werte des Quaders zur explorativen Untersuchung des Verhaltens der Eigenwerte und Faktoren. IP-GIPT. Erlangen: https://www.sgipt.org/wisms/fa/quader/q50.htm
Copyright & Nutzungsrechte
Diese Seite darf von jeder/m in nicht-kommerziellen Verwertungen frei aber nur original bearbeitet und nicht inhaltlich verändert und nur bei vollständiger Angabe der Zitierungs-Quelle benutzt werden. Das direkte, zugriffsaneignende Einbinden in fremde Seiten oder Rahmen ist nicht gestattet. Zitate und Links sind natürlich erwünscht. Sofern die Rechte anderer berührt sind, sind diese dort zu erkunden. Sollten wir die Rechte anderer unberechtigt genutzt haben, bitten wir um Mitteilung. Soweit es um (längere) Zitate aus ... geht, sind die Rechte bei/m ... zu erkunden oder eine Erlaubnis einzuholen.
Ende_ q50_Datenschutz_Überblick _ Relativ Aktuelles _ Rel. Beständiges _ Titelblatt _ Konzept _ Archiv _ Region _ Service iec-verlag _ Mail: _ sekretariat@sgipt.org _
14.05.2015 Linkfehler geprüft und korrigiert, Layout-Anpassung.